Lösungen Kapitel 36
From BioPhy.de Wiki
Contents |
Aufgabe 36.1:
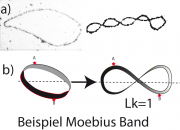
Experimentelle Bestimmung der Superverdrillung.
Die Anleitung zu dieser experimentellen Aufgabe findet man in Kapitel 36.2.1 (Kleindruck). Man benötigt dazu nur ein flaches Band aus Stoff, Besenstiele und Bleistifte verschiedener Dicke. Man muss die Enden des Bandes nach dem Umwickeln festhalten und durch Lockern der Spirale vom die Zylinder entfernen. Die folgende Abbildung zeigt was Sie sehen sollten, wenn Sie Superverdrillungen von Lk=4 und Lk=1 einstellen:
Aufgabe 36.2:
DNA als Kuhn-Kette (SAW-Modell)
Zur Erinnerung an die Struktur der Doppelhelix: Der Durchmesser ist 2 nm. Der Abstand zwischen benachbarten Basenpaaren (bp) ist 0.34 nm und benachbarter Paare sind um ca 36\degree gegeneinander gedreht. Die Ganghöhe ist ca 3.4 nm und die Zahl von Basenpaaren pro Ganghöhe ist 10 bis 10.4. Da die Konturlänge der DNA aus 10 000 bp ist, besteht sie aus
Segmenten der Kuhnlänge
.
Der Flory-Radius in Lösung (dreidimensional):
Der Flory-Radius in Lösung (zweidimensional):
Der ideale Kettenradius wird in 3D:
Messungen der End-zu-End-Abstände (Flory-Radien ) liegen vor allem für auf Oberflächen adsorbierter DNA vor. Es gibt mehrere Methoden dazu, von denen wir zwei betrachten: erstens, AFM Untersuchungen von auf Festkörpern adsorbierter DNA [1, 2] und zweitens, Messungen mittels Mikrofluoreszenz von DNA-Filamenten auf festkörpergestützten Membranen aus DOPC, die durch Dotierung mit DOTAB positiv aufgeladenen wurden [3].
Wir zeigen in Abb. 1 zunächst Ergebnisse von Meier und Rädler [3]. Wie Abb. 1a links und in der Mitte zeigt, schwimmen die Filamente fluiden Membranen, die auf Festkörpern ruhen. Das rechte Bild zeigt den Flory-Radius als Funktion der Zahl N der Basenpaare. Aus der doppelt logarithmischen Auftragung folgt das Skalengesetz
Dies entspricht recht gut dem Skalengesetz zweidimensionaler Makromoleküle, für die der Exponent 0.75 ist. Die RNA aus 48000 bp besteht aus 685 Kuhn-Segmenten. Man erwartet einen End-zu-End-Abstand von . Der in Abb1 Mitte gefundene Wert liegt bei
.
Abb.2 zeigt das Ergebnis der Messung mittels AFM. Das linke Bild zeigt eine Aufnahme eines verkürzten Fragments. Rechts ist der End-zu-End-Abstand gegen die Konturlänge der Filamente aufgetragen, wobei
ist). Man beobachtet zwei Regime. Für
ist der Exponent
und für
wird
. In diesem Experiment verhält sich die adsorbierte DNA wie eine Flory-Kette in Lösung. Man findet also dreidimensionales Verhalten, im Gegensatz zú der Situation in Abb.1. Der Grund ist wahrscheinlich die sehr geringe Adsorption der DNA auf Glimmer.
Die beiden Experimente zeigen, dass die DNA-Filamente interessante Modellsysteme zur Untersuchung fundamentaler Eigenschaften von Makromolekülen ist. Da DNA auch geschlossene Ringe bildet, eignet sich das Molekül auch dazu, das Verhalten exotischer Makromoleküle zu untersuchen [3].
Referenzen:
1 Valle, F., et al.(2001). Scaling Exponents and Probability Distributions of DNA End-to-End Distance. Phys. Rev. Lett. 95: 158105.
2 Meier, B., Rädler, J. (2001) DNA on Fluid Membranes: A Model Polymer in Two Dimensions Macromolecules, 34, 5723-5724
3 Witz et al. (2005) Phys. Rev. Letters 101: 148103
Aufgabe 36.3:
Die Redundanz des genetischen Codes und die Wobble Hypothese
I. Aufgabe des genetischen Codes ist die Kodierung der 20 vorkommenden natürlichen Aminosäuren. Verfügbar ist ein biologisches Alphabet mit den vier Buchstaben A (Adenin), T (Thymin), C (Cytosin) und G (Guanin). Die Zahl Z von Objekte, die bei einer Wortlänge von n Buchstaben (Basen) durch beliebige Kombination von Buchstaben kodiert werden können, ist (Z = Zahl von Kombinationsmöglichkeiten):
Damit Z = 20 wird, muss sein, d.h. die minimale Wortlänge ist
. Damit ließen sich im Prinzip Z = 64 Aminosäuren kodieren. Drei der Codons (die Stop-Codons UAG, UGA, UAA) werden für die Beendigung der Translation benötigt. Eines der Tripletts (am häufigsten AUG) dient als Start-Codon. Die restlichen 61 Tripletts werden benutzt, um manche Aminoisäuren mehrfach zu kodieren. Der genetische Code ist also entartet. Er ist jedoch eindeutig, d.h. es gibt kein Triplett, das verschiedene Aminosäure kodiert. Die Entartung ist nicht gleichmäßig. Serin und Leucine sind 6-fach, Tyrosin und Histidin sind zweifach und Tryptophan und Methionin einfach entartet.
II. Die Entartung ist heterogen, wie Abb.1 zeigt. Nur zwei Aminosäuren werden durch ein Codon repräsentiert. Interessanterweise fungiert das Triplett von Methionin (AUG) gleichzeitig als Startcodon. Glutaminsäure wird durch zwei Tripletts kodiert. Aber nur die dritte Base ist variabel und kann entweder aus A oder G bestehen. Man sagt, sie ist 'zweifach entartet'. Das Codon von Glycin ist nach dieser Nomenklatur vierfach entartet.
III. Die Wobble (oder Wackelbasen-)-Hypothese versucht folgendes Problem zu erklären: Für die Biosynthese (Translation) wird im Prinzip für jede der 61 möglichen Codons eine t-RNA mit dem spezifischen Anticodon benötigt, Die meisten Organismen besitzen aber nur 43 t-RNA. Die Anticodons von einigen der t-RNA müssen also mit mehr als einem Codon paaren. Daher postulierte F. Crick 1966, dass die Bindung der 5'-Base der t-RNA an die 3'-Base der Boten-RNA (m-RNA) schwächer ist als die in den Basenpaaren der DNA. Die Wasserstoffbrückenbindung könnte z. B. lockerer sein. Einige Beispiel findet man in [1].
Referenz:
1 Berg, JM. et al. 'Biochemie', Ch. 29.3.9.
Aufgabe 36.4:
Beweis der Gleichung 36.6 (nicht Gl 36.9b).
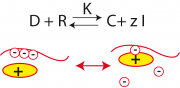
Wir betrachten als Beispiel die Bildung eines Komplexes (C) aus dem Repressor (R) und der DNA (D). Bei dieser Reaktion zweier elektrisch geladener Makromoleküle werden gleichzeitig Gegenionen frei. Die Reaktionsgleichung hat daher die in Abb.1 angegeben Form:
Die Gleichgewichtskonstante der Reaktion ist:
Logarithmieren liefert:
Da die Messung mit einwertigen Salzen durchgeführt wurde, ist gleich der Konzentration
des Salzes, womit Gl. 36.6 begründet ist. Diese Gleichung wurde zunächst empirisch begründet. Die obige Rechnung zeigt, dass das logarithmische Gesetz darauf beruht, dass gleichzeitig mit der Dissoziation des RD-Komplexes z einwertige Gegenionen frei werden.
Aufgabe 36.5:
Visualisierung der Lac-Operator-Repressor Bindung
Vorbemerkung: In Kapitel 36.4 (Abb. 36.6 und 36.7) wurde die Kontrolle der Genexpression in Prokaryonten durch die Wechselwirkung zwischen dem Operon und dem Repressor (R) beschrieben. Der Repressor besteht aus einem Dimeren, das an das Operon bindet und die Expression blockiert, solange der Lactose-Spiegel hoch genug ist. In der DNA finden sich neben dem Operon andere, etwas schwächer bindende Bindungsstellen für R. Durch Bildung von Tetrameren von R können diese Bindungsstellen an den eigentlichen Operator andocken, wodurch weit entfernte Segmente der DNA miteinander wechselwirken können. Dieses Prinzip der Kopplung von Segmenten der DNA, die um mehrer hundert Basenpaare (bp) vom Operon getrennt sein können, spielt eine besonders wichtige Rolle für die Regulation der Genexpression in Eukaryonten durch verschiedene Transkriptionsfaktoren, Repressoren und Verstärkern der Genexpression (s. Kapitel 36 und [1] Ch. 7).
Die ersten Beobachtungen der Kopplung weit entfernter Bindungsstellen durch Repressor-Tetramere wurden mittels EM gemacht. Dabei wurden Konstrukte der DNA hergestellt, in denen zwei Operone in verschiedenen Abstände in DNA eingebaut wurden. Bei Bindung zweier Operone durch R-Teramere bildet die DNA Schleifen, die man im EM beobachten kann, (s.Abb 1a links). Solche Experimente liefern jedoch keine Einblicke in die Dynamik der Schleifenbildung oder die Lebensdauer der O-R-Bindung. Das Problem wurde in dem eleganten Experiment von Finzi und Gellies [3] gelöst (s. Abb. 1b und 1c). Es wurden zwei Konstrukte hergestellt: eines mit zwei Operone im Abstand von 305 bp (Abb. 1b oben links) und eines mit nur einem O (Abb. 1b oben rechts), das zur Kontrolle diente. Wie in Abb. 1a rechts unten gezeigt wurde die DNA mit einem Ende auf einer Glasoberfläche fixiert (über Biotin-Streptavidin Koppler) während an das andere Ende ein Polystyrolkügelchen mit Durchmesser angedockt wurde.

Beobachtet wurde die Brownsche Bewegung der Sonde. Information über die Schleifenbildung liefert die Bewegungsfreiheit die durch die Reichweite der mittleren Verschiebungsquadrats bestimmt ist. Die R-O-Bindung ist dynamisch. Daher beobachtet man statistische Übergänge zwischen einem Zustand mit größerem und einem mit kleinerem Wert von
(siehe Abb.2a). Die Verteilung
der Bewegungsfreiheitist daher bimodal. Die obere Spur der Abb. 1a zeigt die Verteilung
für den Fall des Kontrollexperiments (s. Abb. 2b). Die Verteilung der Lebensdauern der Schleifenbildung ist exponentiell. Sie ist also durch eine Übergangswahrscheinlichkeit bestimmt. Diese ist
.
Referenzen:
1 Alberts B., Johnson A., Walter P., Lewis J., Raff M., Roberts K., (2008) Molecular Biology of the Cell. 5th ed. Taylor and Francis. New York.
2 Krämer, H., et al. (1987), Lac repressor forms loops with linear DNA carrying two suitably spaced lac operators. The EMBO Journal 6:.1481-1491
3 Finzi, L., Gelles, J. (1995) Measurement of lactose-repressor -mediated loop formation and breakdown in single DNA molecules. Science 267:378380.
Aufgabe 36.6:
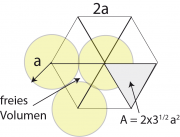
Die maximale Packungsdichte der DNA in Viren.
Abb. 1 zeigt einen Querschnitt durch das Dreiecksgitter. Bei maximaler Packung der DNA ist die Seitenlänge der Dreiecke gleich dem Durchmesser 2a der Filamente. Die mit DNA gefüllte Fläche ist . Die Fläche des Dreiecks ist
. Daher ist der mit DNA besetzte Bruchteil der Fläche:
.
Die gesamte Länge der DNA ist oder
wobei
die Kuhnlänge (
) ist. Der Radius des (als sphärisch angenommenen) Capsids ist
oder
und das Volumen
.
Bei dichter Packung der DNA ist der Abstand zwischen den Filamenten durch folgende Bedingung bestimmt: . Mit
folgt schließlich für den Abstand zwischen den Filamenten
.
Aufgabe 36.7:
Beobachtung der Helixbildung durch Zirkulardichroismus. (CD= circular dichroism).
Helikale Strukturen besitzen stets chirale Symmetrie. Diese kann man auf zwei Weisen beobachten. Erstens durch Messung des Drehvermögens des elastisch gestreuten Lichts und zweitens mittels Cirkulardichroismus (CD-)-Spektroskopie.
Methode I. Linear polarisiertes Licht, das durch eine Küvette der Dicke d tritt wird um einen Winkel gedreht. Ursache ist der unterschiedliche Brechungsindex für rechts (
) und links (
) drehende zirkular polarisierte Wellen. Da das linear polarisierte Licht als Überlagerung einer rechts und einer links drehenden Welle dargestellt werden kann, dreht die Polarisationsrichtung um
.
heißt optische Rotation. Die Phase der Lichtwelle ist generell
. Daher wird
. Man misst in der Regel
als Funktion der Konzentration in
.
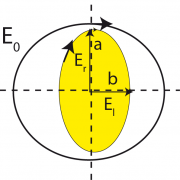
Methode II: Die CD Spektroskopie basiert auf der Messung der unterschiedlichen Absorption (oder Transmission) von links und rechts zirkular polarisiertem Licht mit den molaren Extinktionskoeffizienten und
. Daher wird die linear polarisierte Lichtwelle nach dem Durchtritt durch die Lösung elliptisch polarisiert, da die Amplituden
und
der Teilwellen nun unterschiedlich sind. Die Elliptizität hat die Form:
wobei T die Transmission ist. Um mit molekularen Eigenschaften zu korrelieren, benutzt man das Lambert-Beer-Gesetz in der Form
einführen. Setzt man
und
in die rechte Seite der obigen Gleichung ein und entwickelt die Exponentialfunktion so folgt:
Anwendungen:
1. Die CD-Spektroskopie ist eine Standardmethode zur Untersuchung der thermisch induzierten Helix-Knäul Umwandlung. Abb. 1a zeigt ein Beispiel aus dem Review von Kypr et al [1].
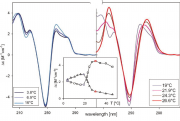
2. Unterscheidung der drei Konformationen A, B, Z der DNA. Die Form B bildet eine Rechtsschraube und ist die Standardform in der Biologie. Die Form Z bildet eine Linksschraube und kommt gelegentlich innerhalb der B-Form vor. In beiden Fällen sind die Basenpaare weitgehend planar. Die A-Form bildet eine Rechtschraube in der die Ebene der Basen gegeneinander verdreht sind wodurch die Helix aufgebläht wird.
Referenzen:
1 Kypr, J. et al. (2009) Circular dichroism and conformational polymorphism of DNA Nucleic Acids Research 37: 1713-1725. Ein frei zugänglicher Artikel mit Beispielen der DNA-Konformationsumwandlung.